GND in lokale Datenbank laden
Normdaten sind hilfreiche und notwendige Datenquellen zum Aufbereiten, Erschließen und Finden von Archivmaterial. In diesem Beitrag laden wir die Gemeinsame Normdatei (GND) zur Weiterverarbeitung in einen lokalen Triple Store von Apache Jena.
 von der
DNB unter [CC BY-SA Lizenz](https://creativecommons.org/licenses/by-sa/4.0/).](/post/2021-06-gnd-in-lokale-datenbank-laden/gnd_hubf082f7bbdaac622ea39cd2aed6726e1_38178_89e8517eb08384fb4e1deb9cb8ec3e07.webp)
DNB unter CC BY-SA Lizenz.
Über die Gemeinsame Normdatei
Die GND ist ein von der Deutschen National Bibliothek (DNB) betreuter Dienst zur kooperativen Nutzung von Normdaten. Im Archivbereich interessieren wir uns insbesondere für eindeutig zu identifizierende Personen, Orte und Dinge.
Die GND liegt im Format MARC 21 und in RDF vor und kann über unterschiedliche Schnittstellen abgerufen werden. Wir konzentrieren uns hier auf die GND Daten im RDF Format, da wir diese effektiver durchsuchen können. Zum heutigen Stand gibt es von Seiten der DNB keinen SPARQL Endpoint für den Zugriff auf die Daten1. Daher spielen wir einen Daten Dump in eine lokale Datenbank ein. Die Verwendung der Daten in einem Memory basierten Graphen z.B. mit RDFlib funktioniert auf Grund der Größe des Graphen nicht.
Datenbank: Apache Jena Fuseki
 von der
Apache® Software Foundation.](/post/2021-06-gnd-in-lokale-datenbank-laden/jena_hu2356985c631d3f3d54e2729962d749aa_49736_9d3ca77f0e108edf462976f508cce2a0.webp)
Apache® Software Foundation.
Als lokale Datenbank für unser Experiment haben wir uns für Apache Jena entschieden. Das Projekt vereint einen Triple Store (TDB) zum Speichern und Verwalten der RDF Triple der GND, eine Implementierung von SPARQL namens ARQ, sowie einen SPARQL Endpoint mit Fuseki. Zusätzlich gib es auch noch Tools zum Arbeiten mit RDF und OWL, die wir erstmal nicht nutzen werden. Da wir gerne mit Docker arbeiten, es aber kein offizielles Docker Image des Apache Projektes gibt, verwenden wir das Docker Image stain/jena-fuseki aus dem Docker Hub2.
Einlesen der Daten
Zum Einlesen haben wir uns die Datei authorities_lds.nt.gz aus dem DNB Data Dump heruntergeladen3.
Wir arbeiten mit der folgenden Ordnerstruktur:
- README.md: Überblick über das Projekt und die Daten geben!
- fuseki: Ordner zum Persistieren der Datenbank von Apache Jena.
- fuseki_test: Ordner für den Test mit Apache Jena.
- gnd_triples: Speicherort der
.nt.gzDatei. - test_triples: Speicherort von Testdateien im
.ttlFormat.
Testdaten einlesen
Als erstes starten wir einen Docker Container mit dem Namen fuseki,
machen die Web GUI verfügbar über http://localhost:8080, setzen
das Administrator Passwort auf 123123 und mounten unsere Testordner in den Container4.
docker run -it --rm --name=fuseki -p 8080:3030 -e ADMIN_PASSWORD=123123 -v "$(PWD)\data\test_triples:/staging" -v "$(PWD)\data\fuseki_test:/fuseki" stain/jena-fuseki
Den Docker Container lassen wir laufen und öffnen ein zweites Terminal (Shell, PowerShell, …).
Von diesem aus können wir uns mit dem Container verbinden und den Einlesevorgang starten.
Hierfür gibt es zwei Varianten. In der ersten wird das bereitgestellte load.sh Skript verwendet, welches auf tdbloader verweist.
Wir verzichten darauf dem load.sh Skript spezifische Dateien vorzugeben, sondern lesen alle kompatiblen Formate im fuseki_test Ordner in die test Datenbank ein.
docker exec -it fuseki /bin/bash -c './load.sh test'
Abschließend wird der geladene Datensatz in der Web GUI angelegt. In dem folgenden Bildschirmfoto in Abbildung 1 wird ein TDB Datensatz mit dem Namen test angelegt. Hier ist der gleiche Bezeichner zu wählen, wie er auch beim Laden verwendet wurde.
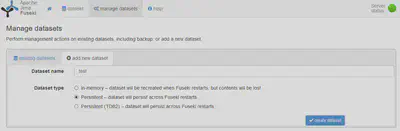
Anschließend kann entweder in der Web GUI im Tab info oder mit dem folgenden SPARQL-Befehl die Anzahl der eingelesenen Trippel bestimmt werden.
SELECT COUNT(*)
WHERE {
?s ?p ?o .
}
Alternative mit TDB2
Alternativ kann auch der tdbloader2 verwendet werden. Hier werden explizit nur Dateien im *.ttl Format eingelesen.
docker exec -it fuseki /bin/bash -c './tdbloader2 --loc=/fuseki/databases/test /staging/*.ttl'
In der Web GUI darauf achten, den Datensatz als TDB2 anzulegen.
GND einlesen
Nach einem erfolgreichen Test lesen wir nun die komplette GND ein.
Wir haben uns für TDB2 entschieden, da wir planen den Datensatz länger zu verwenden. Das Einlesen benötigt mit 6 Stunden und 15 Minuten etwa eine Viertelstunde länger als mit der Vorgängerversion TDB.
1. Starten des Containers mit Daten zum Einlesen
Der folgende Befehl startet einen Docker Container mit dem Verzeichnis der Daten zum Einlesen und einem Verzeichnis zum Speichern der Datenbank von Apache Jena.
docker run -it --rm --name=fuseki -p 8080:3030 -e ADMIN_PASSWORD=123123 -v "$(PWD)\data\gnd_triples:/staging" -v "$(PWD)\data\fuseki:/fuseki" stain/jena-fuseki
2. Einlesen der Daten im TDB2 Format
In einem separaten Terminal starten wir den Einlesevorgang der GND Daten mit dem tdbloader2.
docker exec -it fuseki /bin/bash -c './tdbloader2 --loc=/fuseki/databases/gnd /staging/*.nt.gz'
3. Anlegen des Datensatzes in der Web GUI
Nach dem erfolgreichen Einlesen legen wir den Datensatz in der Weg GUI an.
Hierbei achten wir darauf, den gleichen Bezeichner zu verwenden, wie beim Einlesen und als Format TDB2 zu wählen.
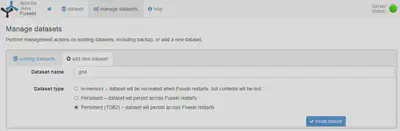
4. Exemplarisches Testen der eingelesenen Daten
Anschließend testen wir exemplarisch, ob die Daten nutzbar sind. Hierfür verwenden wir eine SPARQL Abfrage mit einem Standardbeispiel aus der GND Ontology.
SELECT ?p ?o
WHERE {
<https://d-nb.info/gnd/118514091> ?p ?o
}
Falls zu diesem Zeitpunkt noch keine Ergebnisse angezeigt werden, den Container beenden und wie in Schritt 5 starten.
5. Wiederverwendung des Containers
Um einen neuen Apache Jena Docker Container zu starten und die eingelesenen Daten wiederzuverwenden, kann der folgende Befehl verwendet werden.
docker run -it --rm --name=fuseki -p 8080:3030 -e ADMIN_PASSWORD=123123 -v "$(PWD)\data\fuseki:/fuseki" stain/jena-fuseki
Zusammenfassung und Ausblick
Zu Beginn war es etwas frustrierend, einen funktionierenden Workflow zum Einlesen der Daten in den Docker Container von Apache Jena aufzubauen. Kleine Abweichungen der obigen Beschreibung oder der Reihenfolge sorgen dafür, dass der komplette Einlesevorgang neu gestartet werden muss.
Die Einlesezeit wird sich auf anderen Systemen drastisch unterscheiden. Das liegt unter anderem daran, dass wir bei unseren Tests die Dateien von einem Windows Dateisystem in ein Linux-Dateisystem im Docker Container eingelesen und dann wieder auf das Windows Dateisystem herausgeschrieben haben.
Mit der Kombination aus Apache Jena, Docker und GND haben wir jetzt eine lokale Testdatenbank, mit der wir intensiv mit den Daten arbeiten können, ohne durch Bandbreiten- oder API-Beschränkungen ausgebremst zu werden.
Zum Zeitpunkt unseres Tests gab es mehrere Probleme, die mit stain/jena-docker#50 gefixt werden sollten. ↩︎
Die Daten werden auch im für die Kompression optimierten HDT Format angeboten, jedoch hätten wir dafür Apache Jena für HDT anpassen müssen. ↩︎
Die Sicherheit des Passworts ist hier nicht relevant, da wir die Anwendung lokal auf unserem Rechner testen. ↩︎
